随着分子生物学技术的发展以及其在微生物生态学及环境微生物学领域的广泛应用,促进了以微生物群落基因组及微生物群落转录组为整体研究对象的学科发展,成为多领域研究的热点。
宏基因组(Metagenome)这一名词最早是由Jo Handelsman等于1998年正式提出,泛指环境样品中所有微小生物总的DNA,主要研究对象包括了细菌、真菌、病毒等,以研究特定生境中微生物群落结构、潜在代谢功能、进化关系及对环境因子响应机制为主。相较于一般扩增子测序仅对微生物群落中物种组成及多样性的深入挖掘来说,宏基因组测序理论上可以全面了解样品中近乎全部微生物类群组成情况,同时对于群落基因组成以及潜在的代谢网络也可进行深入的挖掘。
而扩增子测序相较于宏基因组测序项目来说,深入挖掘样品物种组成所需成本相对较低,建议在进行宏基因组项目前,优先采用扩增子测序预估研究对象中的微生物群落组成复杂性(一般微生物宏基因组中以细菌为主,除特定研究真菌宏基因组或病毒宏基因组等),根据物种组成多样性情况来确定采用建库测序方式、测序数据量以及分析流程。
对于宏基因组项目来说,根据研究目的大致可以分为两类。
l 若样品中微生物群落组成较复杂,研究目的基于比较不同样品间整体微生物群落物种及功能组成差异及与理化因素间的相关性分析为主,则对测序数据量的要求不高,采用PE250或PE150,构建小片段文库,高度复杂的群落,拼接的意义不大,重点在于分组间的物种及功能组成的差异比较,一般测序数据量5-10Gb均可。
l 若样品属于极端生境或富集培养等样品,物种组成较为简单,集中体现为占据超高丰度的有限数目优势类群,研究目的主要在于挖掘并恢复其中优势类群的基因组草图信息,特别是未培养类群,则建议加大测序量;条件允许可采用更长读长测序进行宏基因组数据组装及主要类群Binning,而后对Bin出的主要类群进行样品间功能丰度的比较分析等。
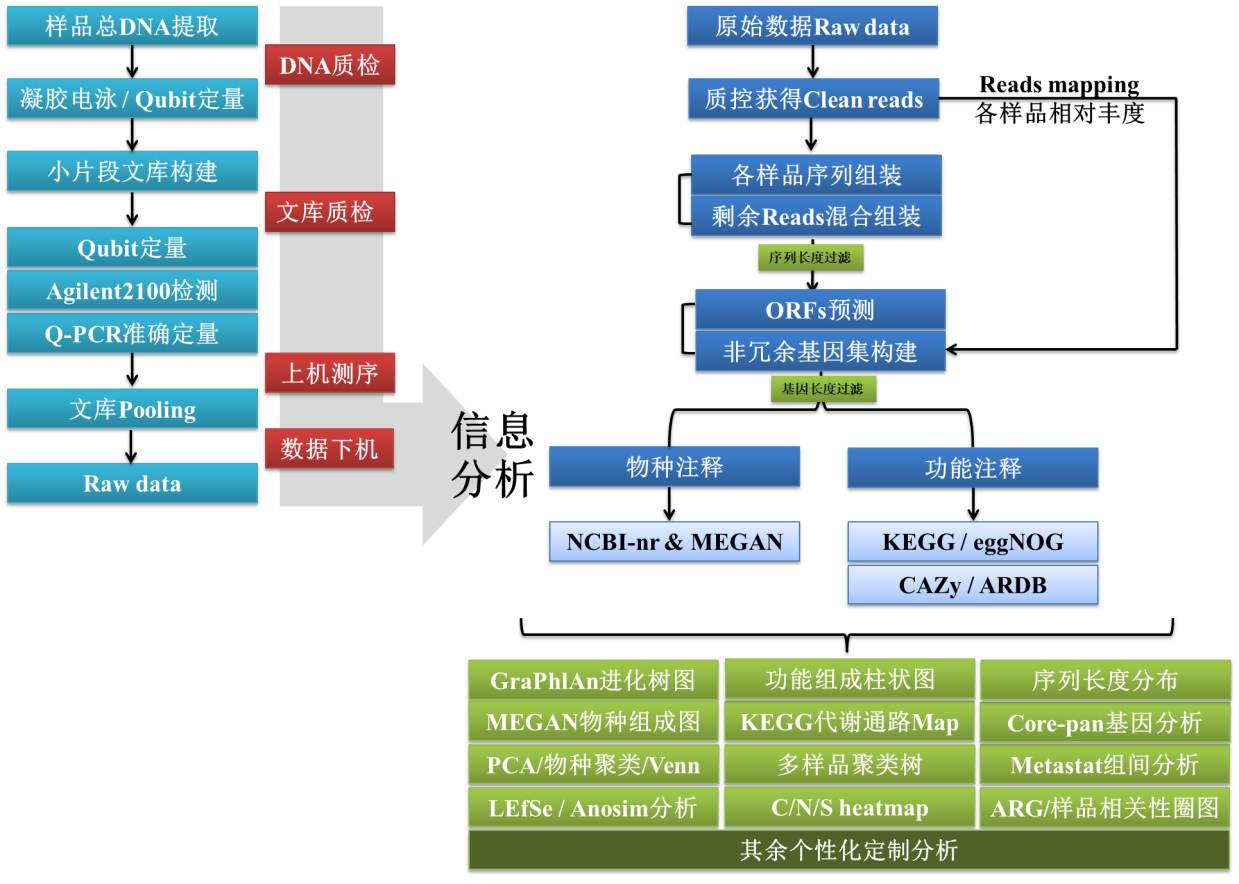
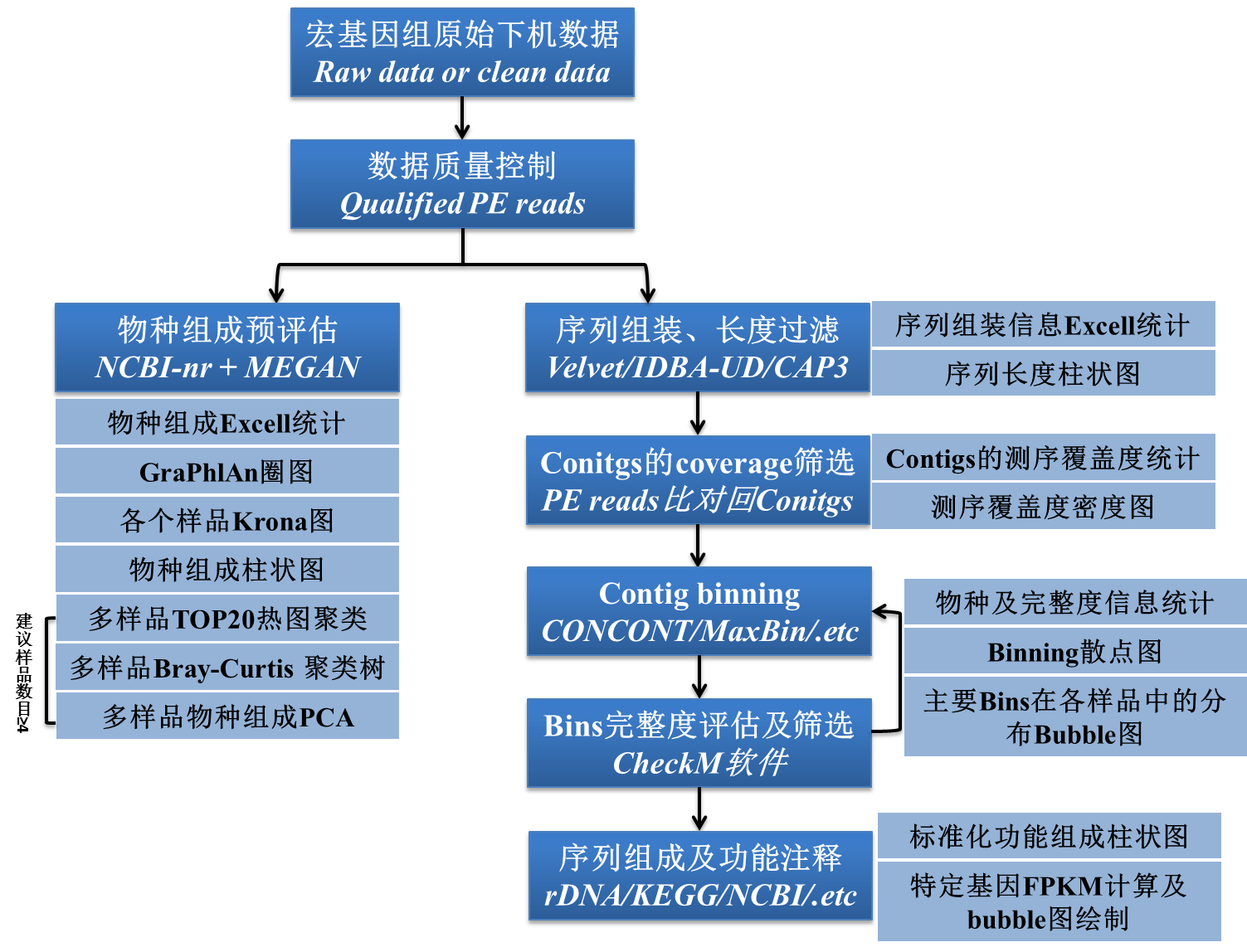
宏基因组项目,相较于扩增子高通量的样品制备,对DNA完整度要求较高,提取到的总DNA电泳图需要有清晰可见的主带,且弥散程度较低,过高的弥散度不利于文库构建。另一方面,随着测序成本的一再降低,宏基因组样品的生物学重复设置也变得基本化。同时,如果同个样品即要做扩增子测序又要做宏基因组测序,建议采用同个DNA,不同DNA可能会产生批次效应。